Abstract
Objective: To isolate and identification Aggregatibacter actinomicetemcomitans from clinical subgingival plaques of periodontitis patients. Methods: Selective media was prepared by adding 200 ug/mL of vancomycin and 10 U/mL of bacitracin, 5% sheep blood to Wilkins Chalgren Anaerobic agar. Subgingival samples collected from patients with moderate to severe periodontitis aged 18- 35 were used to recover Aa. The colony was then identified by the MALDI-TOF method and PCR analysis. Results: The Aa was successfully cultured and stored from a clinical sample of the 12th 25 years-old patient who had moderate aggressive periodontitis. Conclusion: Aa is a major putative oral pathogen and concerned with other systemic diseases. This pure bacterial culture will be helpful for other experimental studies in dental and medical areas.
ĐẶT VẤN ĐỀ
Bệnh lý nha chu bao gồm bệnh lý nướu và viêm nha chu tác động hơn 90% dân số trên toàn thế giới 1 , phá huỷ mô nâng đỡ răng và là nguyên nhân gây mất răng sớm. Nguyên nhân phát triển các bệnh lý này là mảng bám răng, trong đó các vi khuẩn bệnh nguyên như Porphyromonas gingivalis (Pg) , Tannerella forsythia (Tf) , Treponema denticola (Td ) và Aggregatibacter actinomycetemcomitans (Aa) được chứng minh là có vai trò quan trọng 2 . Hoạt động của mảng bám vi khuẩn, đáp ứng của vật chủ và các yếu tố nguy cơ toàn thân và tại chủ là các thành phần tham gia trong sinh bệnh học các bệnh lý nha chu. Hoạt động của bệnh nha chu có thể từ chậm, mạn tính, phá huỷ mô từ từ đến bùng phát cấp tính với cường độ và thời gian thay đổi khác nhau về mức độ nghiêm trọng. Aa được cho là bệnh nguyên có liên quan tới viêm nha chu tiến triển ở các bệnh nhân trẻ tuổi với hình thức bệnh liên quan tới nhiều răng với tốc độ mất mô nha chu nhanh, đáng kể, tuổi khởi phát sớm 3 .
Aa có tên gọi trước đây là Actinobacillus actinomycetemcomitans , là một vi khuẩn gram âm, kích thước 0,5–0,8μm×0,6–1,4 μm, kị khí tuỳ nghi 4 . Về độc lực, leukotoxin của Aa là một trong những độc tố mạnh nhất, có khả năng ức chế trực tiếp các yếu tố miễn dịch của con người như bạch cầu đa nhân, immunoglobulin và hệ thống bổ thể. Tương tự như các vi khuẩn bệnh nguyên khác, Aa bám dính vào mô vật chủ và các vi khuẩn khác bằng hệ thống lông, tiết ra invasin để xâm nhập vào các đại thực bào và các enzim để phá huỷ mạng lưới sợi collagen (collagenase). Aa cũng ức chế sự sản xuất các immunoglobulin (Ig) G và M và tăng sinh nguyên bào sợi, có nội độc tố (lipopolysaccharide-LPS) gây viêm và tiêu xương 5 . Bên cạnh việc có mặt trong các bệnh lý trong khoang miệng, vi khuẩn này còn được phân lập trong một số trường hợp viêm nội tâm mạc, áp xe não hay áp xe dưới da 6 , bệnh phổi 7 , bệnh lý mạch vành 8 , đái tháo đường, Alzheimer và viêm khớp dạng thấp 9 .
Nuôi cấy vi khuẩn Aa cần sử dụng môi trường chọn lọc và vi khuẩn phát triển tốt trong môi trường vi hiếu khí với 5-10% CO 2 , ở nhiệt độ 37º C. Trên thạch nuôi cấy, Aa hình thành các khuẩn lạc nhỏ, màu trắng, với đường kính khoảng 0,5–1,0 mm 4 . Nhiều môi trường chọn lọc khác nhau cho Aa đã được phát triển cho các bệnh phẩm lâm sàng, hỗ trợ sự phát triển của một số chủng Aa , với sáu kiểu huyết thanh từ a – f 10 .
Để định danh vi khuẩn Aa , bên cạnh việc dựa trên hình dáng và màu sắc đặc trưng của quần thể, người ta tiến hành nhuộm gram, phân tích sinh hoá, và sử dụng các phương pháp hiện đại hơn như PCR (Polymerase chain reaction), MALDI-TOF (Matrix-assisted laser desorption ionization-Time of flight). Theo Xuedong Zhou và cs (2006), phương pháp sinh hoá đòi hỏi các thử nghiệm trên môi trường nuôi cấy lỏng, do đó đối với một số vi khuẩn có khả năng sinh trưởng thấp như Aa có thể cho kết quả không chính xác 4 . Trong những năm gần đây, sinh học phân tử phát triển mạnh mẽ và được sử dụng để nhận dạng các chủng vi khuẩn khác nhau. MALDI-TOF là phương pháp ion hóa mẫu hấp thụ dựa trên sự hỗ trợ của các chất nền và năng lượng laser. Công nghệ khối phổ giúp định danh vi khuẩn chính xác và thời gian đưa ra kết quả nhanh, bảo quản kết quả và chia sẻ thông tin kỹ thuật số dễ dàng hơn 11 . Đối với Aa , theo tác giả Hussain và cs (2020), việc định danh nhanh chóng vi khuẩn này trong mẫu mảng bám dưới nướu chính xác chỉ trong thời gian 2 phút với kỹ thuật MALDI-TOF 12 . Real-time PCR (Polymerase chain reaction) hay phản ứng chuỗi trùng hợp là một phương pháp đơn giản sao chép trình tự DNA hay RNA, được ứng dụng để phát hiện cả các vi khuẩn bệnh nguyên nha chu. Phương pháp này không đòi hỏi tế bào sống nhưng có khả năng nhận biết nhiều loại vi khuẩn bệnh nguyên khác nhau với độ nhạy cao hơn có ý nghĩa so với nuôi cấy vi khuẩn. Đối với Aa , các nghiên cứu sử dụng công nghệ real-time PCR cho kết quả tương đương hoặc cao hơn với nuôi cấy thông thường đồng thời nhanh và nhạy hơn trong việc phát hiện Aa ở mảng bám dưới nướu 13 .
Qua nhiều nghiên cứu, Aa có mặt trong mảng bám dưới nướu ở cả bệnh nhân khoẻ mạnh và viêm nha chu, đặc biệt vai trò quan trọng trong sinh bệnh học của viêm nha chu tiến triển 9 , 14 , 15 . Vi khuẩn nuôi cấy trực tiếp từ bệnh nhân lâm sàng cũng được cho có độc lực mạnh hơn và có giá trị trong các thử nghiệm in vitro 16 . Do đó, mục tiêu của nghiên cứu này là phân lập vi khuẩn Aa từ mảng bám dưới nướu của bệnh nhân viêm nha chu trung bình tới nặng, sử dụng phương pháp MALDI-TOF và giải trình tự để định danh, từ đó lưu trữ mẫu vi khuẩn cho các thử nghiệm về vi khuẩn nha chu.
ĐỐI TƯỢNG VÀ PHƯƠNG PHÁP NGHIÊN CỨU
Đối tượng nghiên cứu
Mẫu mảng bám dưới nướu của bệnh nhân viêm nha chu
Tiêu chuẩn chọn mẫu
Bệnh nhân viêm nha chu mức độ trung bình tới nặng theo tiêu chuẩn phân loại của Hiệp hội nha chu Hoa Kỳ năm 2015, độ tuổi từ 18 tới 35 17 .
Tiêu chuẩn loại trừ
Các đối tượng không sử dụng thuốc kháng sinh, kháng viêm, thuốc tránh thai trước khi tham gia nghiên cứu 6 tháng, không có thói quen hút thuốc lá.
Phương pháp nghiên cứu
Nghiên cứu in vitro
Quy trình nghiên cứu
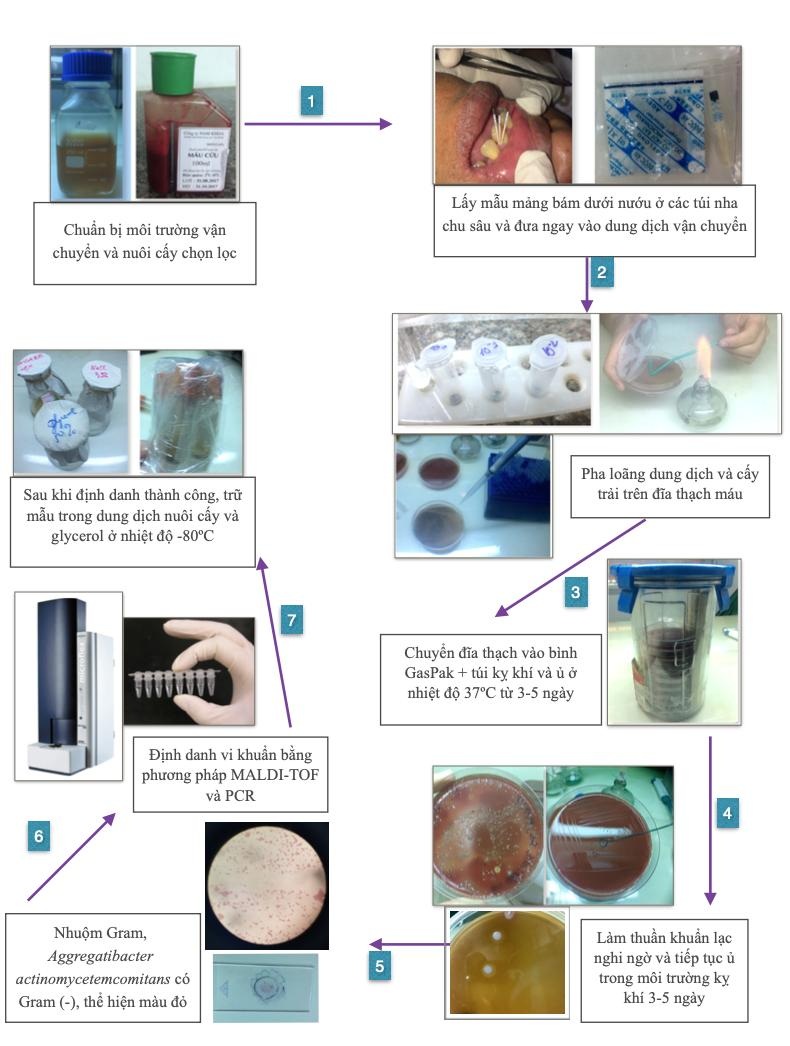
Lấy mẫu lâm sàng : Quy trình lấy mẫu được thực hiện theo phương pháp của Doan Nguyen và cs (2012) 18 . Cô lập vùng răng lấy mẫu bằng gòn cuộn, làm sạch mảng bám trên nướu, sử dụng 2-4 côn giấy vô trùng kích thước 35-40 đưa vào các túi nha chu từ 5mm trở lên, giữ trong 30s, và chuyển vào các ống eppendorf chứa môi trường vận chuyển, đưa vào túi chứa gói hút oxy. Môi trường nuôi cấy chọn lọc : 2ml Wilkins Chalgren Anaerobic broth base (Oxoid- CM0643B) có thành phần hemin và menadion đã hấp vào ống eppendorf để vận chuyển. Môi trường nuôi cấy bao gồm hai loại thạch: loại thứ nhất gồm Wilkins Chalgren Anaerobic broth base (Oxoid- CM0643B và Agar bacteriological-Oxoid LP0011B). Sau khi hấp, bổ sung 5% máu cừu tách sợi (Công ty Nam Khoa), bacitracin (10,000 U/l-Oxoid) và vancomycin (9ug/ml-Oxoid) và đổ 15ml môi trường vào từng đĩa petri 10 , 19 . Loại thạch môi trường thứ hai tương tự nhưng thay máu cừu bằng serum nhau thai bò (Sigma-F7524).
Nuôi cấy và định danh: Vortex mẫu trong 45s, phần huyền phù vi khuẩn được pha loãng bậc 10 trong dung dịch vận chuyển, sử dụng que trải cấy dung dịch mẫu vào đĩa thạch. Ủ ở 37ºC từ 3-5 ngày trong bình Gaspak (Merk-Hoa Kỳ) với túi tạo môi trường kị khí AnaeroPack® (Mitsubishi, Nhật Bản). Khuẩn lạc có kiểu hình và màu sắc đặc trưng được cấy chuyền, làm thuần, định danh bằng phương pháp MALDI-TOF và phương pháp PCR để giải trình tự với cặp mồi đặc hiệu: mồi xuôi (ChDC-Aa-F), 5' - TAG CCC TGG TGC CCG AAG C-3'; mồi ngược (ChDC-Aa-R), 5' - CAT CGC TGG TTG GTT ACC CTC TG-3'20. Quy trình giải trình tự Sanger được thực hiện bởi hãng First Base (Malaysia) bằng máy ABI Genetic Analyzers 3730 XL. Sau đó, chủng vi khuẩn được lưu trữ trong môi trường chứa glycerol ở điều kiện âm sâu trong N2 lỏng ( Figure 1 ). Chủng vi khuẩn được hoạt hóa và cấy chuyền mỗi nửa tháng để đánh giá sự ổn định của chủng trong lưu trữ. Chủng được cho là có khả năng tăng sinh và phục hồi khi chủng nuôi cấy có thể phát triển được trên bề mặt thạch sau nhiều lần cấy chuyền.
KẾT QUẢ
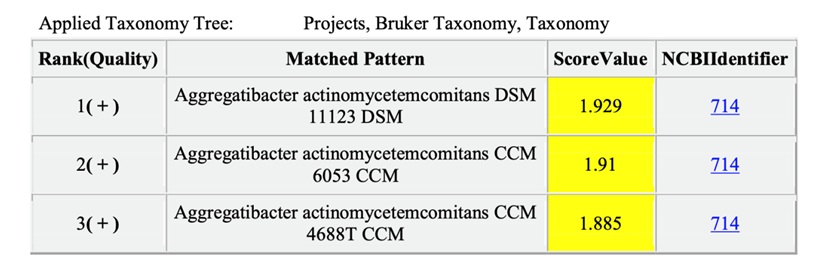
Từ 27 mẫu mảng bám, chúng tôi đã phân lập được 1 chủng vi khuẩn Aa . Khuẩn lạc có hình tròn, có vân màu trắng như ngôi sao, thể lồi, bờ không đều, không tiêu huyết, kích thước khoảng 0,5-1mm ( Figure 2 ). Khi cấy chuyền qua đĩa thạch không chứa máu cừu, khuẩn lạc đôi khi biểu hiện thành hình tròn trơn láng có bờ đồng nhất hơn ( Figure 3 ). Sau khi làm thuần, mẫu khuẩn lạc được gửi định danh bằng phương pháp Maldi-Tof ( Figure 4 ). Kết quả khẳng định vi khuẩn Aa cần tìm. So sánh kết quả gửi giải trình tự bằng phương pháp Sanger với đoạn mồi đặc hiệu 20 với công cụ tìm kiếm các trình tự tương đồng (https://www.ncbi.nlm.nih.gov/BLAST/) khẳng định chủng phân lập thuộc loài Aa ( Figure 5 ).
Figure 2 . (Bên trái) hệ vi khuẩn nuôi cấy từ mảng bám dưới nướu; (bên phải) vi khuẩn có hình dạng và màu sắc nghi ngờ Aa được làm thuần trên đĩa thạch máu
Figure 3 . Khuẩn lạc Aa được cấy trải (bên trái) khi mới làm thuẩn và hoạt hoá lần thứ 24 sau 12 tháng (bên phải) trên đĩa thạch không bổ sung máu cừu cho thấy hình dạng có hơi thay đổi, tuy nhiên vi khuẩn vẫn phát triển bình thường
Figure 5 . Kết quả giải trình tự theo phương pháp Sanger 16S với đoạn mồi đặc hiệu (trên) và so sánh kết quả với ngân hàng gen (dưới); M: DNA molecular weight maker (HyperLadder TM 100bp)
THẢO LUẬN
Aa được chứng minh là có liên quan đến bệnh viêm nha chu ở những người trẻ tuổi 6 , 21 , 22 . Aa có mặt ở 90% viêm nha chu tiến triển khu trú và 30-50% trong viêm nha chu mức độ nặng ở người lớn 15 . Bên cạnh đó, các nghiên cứu dịch tễ và thực nghiệm đã cho thấy Aa cũng được phân lập ở bệnh nhân khoẻ mạnh 21 và vi khuẩn này có liên quan tới các bệnh lý toàn thân như mạch vành, viêm nội tâm mạc, viêm khớp dạng thấp và áp xe não 7 , 8 , 9 . Nếu việc phát hiện và định lượng vi khuẩn Aa hiện nay đã dần được thay thế bằng các phương pháp hiện đại như PCR, nuôi cấy vi khuẩn vẫn giữ vai trò thiết yếu trong việc cung cấp nguồn vi khuẩn để thực hiện các nghiên cứu về đặc điểm miễn dịch và tính chất enzyme, về phương pháp nuôi cấy 19 , 23 hay thử nghiệm hoạt chất điều trị 24 . So với các vi khuẩn hiếu khí, các vi khuẩn kị khí tuỳ nghi như Aa sinh trưởng tốt trong môi trường vi hiếu khí ở nhiệt độ tối ưu (37º C). Thời gian nuôi cấy của Aa thay đổi trong các nghiên cứu khác nhau, từ 24-48 h 25 , 72 h 10 , 26 , 5 ngày 19 , 7-10 ngày 27 , phụ thuộc và phương pháp lấy mẫu và môi trường nuôi cấy. Trong nghiên cứu của chúng tôi, Aa bắt đầu xuất hiện trên đĩa khuẩn lạc khoảng 4 ngày. Có nhiều môi trường nuôi cấy dành cho Aa được tìm thấy trong y văn. Trong nghiên cứu này, chúng tôi sử dụng môi trường nuôi cấy chọn lọc được đề xuất bởi Tsuzubaki và cs (2008). Thành phần của môi trường nuôi cấy này bao gồm các chất dinh dưỡng thiết yếu (như nấm men, peptone, L-arginine, casein và các muối), hemin và menadion, và kháng sinh 10 . Vai trò của kháng sinh bacitracin là ức chế một số lượng lớn các vi sinh vật trong miệng 25 . Vancomycin có tác dụng loại bỏ sự phát triển của các loài thuộc chi Streptococcus và các vi khuẩn gram dương 26 . Theo Tsuzubaki, môi trường chọn lọc được bổ sung 300 lg (khoảng 20 U) bacitracin / mL không thể phân lập Aa vì nó không thể ngăn chặn sự nhiễm của liên cầu khuẩn, các loài thuộc các chi Haemophilui , Eikenella và Neisseriae 10 . Các chủng Aa đều có khả năng kháng với nồng độ cao bất thường của bacitracin và vancomycin. Nồng độ ức chế tối thiểu của bacitracin và vancomycin đối với Aa lần lượt là 400 U / mL và 400 lg / mL. Tuy nhiên, các loại kháng sinh có nồng độ thấp có khả năng chọn lọc và hỗ trợ phát triển sẽ được ưu tiên hơn 25 . Trong nghiên cứu của chúng tôi sử dụng hai loại thạch môi trường chọn lọc, chứa máu cừu hoặc chứa huyết thanh nhau thai bò. Aa đều phát triển được trên cả hai môi trường. Theo Slots và cs (1982), việc thay máu cừu bằng huyết thanh bò không ảnh hưởng tới tỉ lệ nuôi cấy vi khuẩn Aa 23 . Vì là vi khuẩn kị khí tuỳ nghi, Aa đòi hỏi môi trường không hoặc rất ít oxy để phát triển. Đó là lý do vì sao chúng tôi sử dụng hệ thống ủ kị khí với bình Gaspak (Merk) và túi tạo môi trường kị khí cho mỗi lần ủ vi khuẩn và túi tạo môi trường kị khí AnaeroPack (Mitsubishi Gas Chemical Co-Japan) 18 .
Mặc dù Aa được cho là có tỉ lệ cao ở bệnh nhân viêm nha chu thanh thiếu niên, tỉ lệ nuôi cấy thành công Aa trong y văn so với các phương pháp khác khá thấp. Trong nghiên cứu của Lau và cs (2004), tỉ lệ nuôi cấy thành công vi khuẩn Aa ở bệnh nhân viêm nha chu là 6,3% 13 . Có lẽ đây là lý do mà tỉ lệ phân lập Aa trong nghiên cứu của chúng tôi cũng rất thấp (3,7%). Một nghiên cứu của Kotsilkov và cs (2015) thực hiện so sánh giữa phương pháp định danh bằng PCR và nuôi cấy trên 300 mẫu mảng bám dưới nướu ở túi nha chu sâu 7mm trở lên, tỉ lệ Aa phát hiện bởi phương pháp giải trình từ là 30%, trong khi đó tỉ lệ nuôi cấy là 0% 27 . Về việc phân bố vi khuẩn Aa trong miệng bệnh nhân viêm nha chu thanh thiếu niên, Muller và cs (2001) báo cáo rằng Aa có thể được phân lập từ bề mặt niêm mạc miệng, lưỡi, nướu bọt và mảng bám dưới nướu, trong đó tỉ lệ Aa tập trung nhiều nhất ở răng cối lớn thứ ba hàm trên và hàm dưới, sau đó là răng cửa bên hàm trên 28 . Bên cạnh đó, do Aa có liên quan mật thiết với viêm nha chu tiến triển ở bệnh nhân trẻ tuổi 17 , mẫu nghiên cứu cũng tập trung ở bệnh nhân viêm nha chu độ tuổi 18-35 với mẫu mảng bám được lấy ở các túi nha chu có độ sâu 5mm trở lên.
Trên bề mặt đĩa thạch, khuẩn lạc Aa có dạng hình tròn, lồi, gồ ghề, bờ không đều và có vân hình ngôi sao. Điều này tương đồng với các miêu tả trong y văn 4 , 6 . Theo Mytherayi và cs (2019), việc cấy chuyền đôi khi dẫn tới một sự thay đổi tự phát từ hình thái khuẩn lạc thô ráp đến trơn láng. Hình thái khuẩn lạc thô ráp và sự kết dính chặt chẽ được cho là do sự hiện diện của các sợi fimbriae (lông) dài và bó trên bề mặt tế bào. Các khuẩn lạc của các biến thể trơn thiếu cấu trúc bên trong giống như cấu trúc bên trong và các tế bào không biểu hiện fimbria. Các biến thể khuẩn lạc nhẵn không có cấu trúc lông phát triển thành các khuẩn lạc lớn, hình tròn mờ đục trên bề mặt thạch 14 .
MALDI-TOF là phương pháp định danh vi khuẩn nuôi cấy dựa trên proteomic nhanh, tin cậy và có giá thành hợp lý. Tuy nhiên, nó có giới hạn bởi chất lượng phần mềm phân tích 11 . Phương pháp MALDI-TOF có tầm quan trọng đặc biệt đối với việc xác định thông thường các mầm bệnh cần thời gian ủ lâu dài để phân lập và không hoạt hóa sinh học như các vi khuẩn kị khí trong khoang. Thiết kế này giúp tinh giản việc xác định các vi khuẩn từ bệnh nhân mắc bệnh lý nha chu. Trong nghiên cứu này, chúng tôi gửi mẫu vi khuẩn phân tích với hệ thống Bruker BioTyper và nhận giá trị log cao nhất là 1,929. Theo hướng dẫn của hệ thống, giá trị log (điểm) này có thể được coi là một xác suất tốt để nhận dạng vi khuẩn thử nghiệm ở cấp độ loài (p<0,05). Hơn nữa, kết quả giải trình tự cho thấy rằng, chủng phân lập thuộc loài Aa . Để chủng phân lập trở thành đối tượng cho các nghiên cứu về cơ chế độc lực hoặc tính kháng khuẩn của chế phẩm y sinh thì chủng phân lập phải tăng sinh bình thường sau thời gian được lưu trữ. Kết quả lưu trữ và tăng sinh chủng Aa theo thời gian đã cho thấy đến thời điểm hiện tại, chủng này đã được hoạt hóa và cấy chuyền sau 12 tháng (mỗi nửa tháng 1 lần), kết quả cấy chuyền F1, F2, F3 vẫn phát triển bình thường.
KẾT LUẬN
Nghiên cứu đầu tiên đã phân lập và lưu trữ thành công chủng vi khuẩn Aa từ mảng bám của bệnh nhân có bệnh nha chu tại Viêt Nam. Nghiên cứu góp phần tạo nguồn vi khuẩn sẵn có phục vụ cho các mục tiêu nghiên cứu về nha chu học và nghiên cứu hoạt tính kháng khuẩn của các chế phẩm sinh học mới.
LỜI CÁM ƠN
Nhóm nghiên cứu chân thành cám ơn TS. Nguyễn Văn Minh Hoàng, Trung tâm nghiên cứu lâm sàng đại học Oxford (OUCRU) tại Bệnh viện Nhiệt đới, TPHCM đã hỗ trợ thực hiện phương pháp Maldi-Tof. Nghiên cứu này được hỗ trợ kinh phí từ Đại học Quốc gia TP. Hồ Chí Minh theo quyết định số 107/QĐ-ĐHQG, ngày 4/2/2021 (Mã số B2021-44-01).
DANH MỤC TỪ VIẾT TẮT
Aa: Aggregatibacter actinomicetemcomitans
Cs: Cộng sự
LPS: Lipopolysaccaride
Maldi-Tof : Matrix assisted laser desorption ionisation - Time of flight (Phương pháp ion hoá mẫu hấp thụ dựa trên hỗ trợ của chất nền và năng lượng laser - Thời gian bay)
Pg : Porphyromonas gingivalis
PCR : Polymerase chain reaction (Phản ứng chuỗi trùng hợp)
Tf: Tannerella forsythia
Td: Treponema denticola
XUNG ĐỘT LỢI ÍCH
Nhóm nghiên cứu cam kết không mâu thuẫn quyền lợi và nghĩa vụ của các thành viên trong nhóm tác giả.
ĐÓNG GÓP CỦA TÁC GIẢ
Phạm Anh Vũ Thụy lên ý tưởng và thiết kế nghiên cứu, hướng dẫn thu thập mẫu, sửa chữa bản thảo của bài báo.
Trần Thị Phương Thảo tham gia thiết kế nghiên cứu, thực hiện việc thu thập và nuôi cấy vi khuẩn; viết bản thảo của bài báo
Nguyễn Thị Thảo Vân: Khám sàng lọc bệnh nhân viêm nha chu thỏa tiêu chí chọn mẫu, lấy mẫu mảng bám vi khuẩn
Lương Thị Mỹ Ngân giám sát và điều chỉnh quy trình nuôi cấy, tư vấn phương pháp định danh vi khuẩn
References
- Pihlstrom BL, Michalowicz BS, Johnson NW. Periodontal diseases. Lancet 2005;366:1809-1820. . ;:. Google Scholar
- Socransky SS, Haffajee AD. Periodontal microbial ecology. Periodontol 2000, 2005;38:135-187. . ;:. PubMed Google Scholar
- Kesic ́L, Petrovic ́M, Obradovic ́R, Pejcic ́A.Theimportance of Aggregatibacter actinomycetemcomitans in etiology of periodontal disease-Mini Review. Acta Medica Medianae 2009;48:35-37. . ;:. Google Scholar
- Xuedong Z, Yuqing L. Atlas of oral microbiology: from healthy microflora to disease (1 ed.). USA: Academic Press, 2015. . ;:. Google Scholar
- Wolf HF, Hassel TM, Color atlas of dental hygiene- Periodontology, USA: Thieme, 2006. . ;:. Google Scholar
- Samaranayake L, Essential of Microbiology for dentistry, 4 ed, Elsevier, 2012. . ;:. Google Scholar
- Storms I, van den Brand M, Schneeberger P, van 't Hullenaar N. Aggregatibacter actinomycetemcomitans pneumonia with chest and abdominal wall involvement. BMJ Case Rep. 2017 Apr 21;2017:bcr2016217377. doi: 10.1136/bcr-2016-217377. PMID: 28432161; PMCID: PMC5534835. . ;:. PubMed Google Scholar
- Pietiäinen M, Kopra KAE, Vuorenkoski J, Salminen A, Paju S, Mäntylä P, Buhlin K, Liljestrand JM, Nieminen MS, Sinisalo J, Hyvärinen K, Pussinen PJ. Aggregatibacter actinomycetemcomitans serotypes associate with periodontal and coronary artery disease status. Journal of Clinical Periodontolology, 2018 Apr;45(4):413-421. . ;:. PubMed Google Scholar
- Oscarsson J, Claesson R, Lindholm M, Höglund Åberg C, & Johansson A. Tools of Aggregatibacter actinomycetemcomitans to Evade the Host Response. Journal of clinical medicine, 2019; 8(7), 1079. . ;:. PubMed Google Scholar
- Tsuzukibashi O, Takada K, Saito M, Kimura C, Yoshikawa T, Makimura M, Hirasawa M. A novel selective medium for isolation of Aggregatibacter (Actinobacillus) actinomycetemcomitans. Journal of Periodontal Research, 2008 Oct;43(5):544-8. doi: 10.1111/j.1600-0765.2007.01074.x. Epub 2008 Jul 4. . ;:. PubMed Google Scholar
- Clark A.E.. Matrix-Assisted Laser Desorption Ionization-Time of Flight Mass Spectrometry: a Fundamental Shift in the Routine Practice of Clinical Microbiology. Clinical Microbiology Reviews,2013; 26(3), 547-603. . ;:. PubMed Google Scholar
- Hussain M.. MALDI-TOF vs. VITEK 2 for identification of Aggregatibacter actinomycetemcomitans chest wall abscess. IDCases,2020; 20, e00749. . ;:. PubMed Google Scholar
- L. Lau. Quantitative real-time polymerase chain reaction versus culture: a comparison between two methods for the detection and quantification of Actinobacillus actinomycetemcomitans, Porphyromonas gingivalis and Tannerella forsythensis in subgingival plaque samples. Journal of Clinical Periodontology 2004; 31, 1061-1069. . ;:. PubMed Google Scholar
- Mythireyi D, Krishnababa MG, Aggregatibacter Actinomycetemcomitans, an Aggressive Oral Bacteria - A Review, International Journal of Health Sciences & Research, August 2012; 2(5). . ;:. Google Scholar
- Raja M.. Aggregatibacter actinomycetemcomitans - a tooth killer?. Journal of clinical and diagnostic research : JCDR, 2014; 8(8), ZE13-ZE16. . ;:. PubMed Google Scholar
- Ansari MA, Butt TM "Effects of successive subculturing on stability, virulence, conidial yield, germination and shelf-life of entomo-pathogenic fungi", Journal of Applied Microbiology,2011; 110, pp. 1460-1469. . ;:. PubMed Google Scholar
- Caton J, Armitage G, Berglundh T, et al. A new classification scheme for periodontal and peri‐implant diseases and conditions - Introduction and key changes from the 1999 classification. J Clin Periodontol. 2018; 45( Suppl 20): S1- S. . ;:. PubMed Google Scholar
- Doan N, Contreras A, Flynn J, et al. "Proficiencies of three anaerobic culture systems for recovering periodontal pathogenic bacteria", Journal of Clinical Microbiology, 1999; 37, pp. 171-174. . ;:. PubMed Google Scholar
- Rurenga P, Raangs E, Singadji Z, Wekema-Mulder G, Veloo AC, van Winkelhoff AJ. Evaluation of three selective media for isolation of Aggregatibacter actinomycetemcomitans. J Periodontal Res. 2013 Oct;48(5):549-52. . ;:. PubMed Google Scholar
- Kim SG, Kim SH, Kim MK, Kim HS, Kook JK. Identification of Actinobacillus actinomycetemcomitans using species-specific 16S rDNA primers. J Microbiol. 2005 Apr;43(2):209-12. . ;:. PubMed Google Scholar
- Wang X.. Prevalence and distribution of Aggregatibacter actinomycetemcomitans and its cdtB gene in subgingival plaque of Chinese periodontitis patients. BMC oral health,2014; 14, 37. . ;:. PubMed Google Scholar
- Gholizadeh P, Pormohammad A, Eslami H, Shokouhi B, Fakhrzadeh V, Kafil HS. Oral pathogenesis of Aggregatibacter actinomycetemcomitans. Microb Pathog. 2017 Dec;113:303-311. . ;:. PubMed Google Scholar
- Slots J. Selective medium for isolation of Actinobacillus actinomycetemcomitans. Journal of Clinical Microbiology. 1982 Apr;15(4):606-9. doi: 10.1128/JCM.15.4.606-609.1982. . ;:. PubMed Google Scholar
- Yang LC, Hu SW, Yan M, et al. "Antimicrobial activity of platelet-rich plasma and other plasma preparations against periodontal pathogens", Journal of Periodontology,2015; 86, 310-318. . ;:. PubMed Google Scholar
- Mandell RL, Socransky SS, A Selective Medium for Actinobacillus actinomycetemcomitans and the Incidence of the Organism in Juvenile Periodontitis, Journal of Periodontology,1981, 51 (10), 593-598. . ;:. PubMed Google Scholar
- Alsina M, Olle E, Frias J. Improved, low-cost selective culture medium for Actinobacillus actinomycetemcomitans. J Clin Microbiol. 2001 Feb;39(2):509-13. doi: 10.1128/JCM.39.2.509-513.2001. . ;:. PubMed Google Scholar
- Kotsilkov K, Popova C, Boyanova L, Setchanova L & Mitov I, Comparison of culture method and real-time PCR for detection of putative periodontopathogenic bacteria in deep periodontal pockets, Biotechnology & Biotechnological Equipment, 2015; 29:5, 996-1002. . ;:. Google Scholar
- Müller HP, Heinecke A, Fuhrmann A, Eger T, Zöller L. Intraoral distribution of Actinobacillus actinomycetemcomitans in young adults with minimal periodontal disease. Journal of Periodontal Research; 2001 Apr;36(2):114-23. . ;:. PubMed Google Scholar

 Open Access
Open Access